ヒストグラムデータの作成¶
イベントデータからTOFヒストグラムの作成¶
検出器ピクセルごとにTOFスペクトルが作成される。実験中に自動的に実行される。
TOFヒストグラムのマージ¶
検出器ピクセルごとにTOFスペクトルを、マージ(合算)する。ピクセルの位置や散乱角が異なるため、TOFを波長に変換した上でマージを行う。 実験中に自動的に作成され、NeXus形式のファイルとして保存される。
S(Q) 化のための補正¶
nvaSq¶
S(Q) 化のための補正はnvaSq.pyを用いて行う。試料形状(円筒、平板)に応じて異なる処理を行う。
円筒形状の試料の補正¶
nvaSqに含まれるnvaSq.basicを用いる。
import nvaSq
d90 = nvaSq.basic("./", "10708.rn", "SiO2glass2.in", "90_1steps")
- 引数説明:
作業ディレクトリ
sample info file名:<例> SiO2glass2.in
run number file : <例> 10708.rn
ヒストグラムデータ:<例> 90_10steps
補正に必要な試料、試料セル、標準試料等のrun numberは、run number file(例では、10708.rn)中で指定されたものを使う ヒストグラムデータは、バンクを指定し、全ピクセルをマージしたデータを使う場合は1steps、10分割したデータを使う場合は10stepsをしている。 データディレクトリに<run number>_90_10steps.nxが存在しない場合にはエラにより停止する。
平板形状の試料の補正¶
nvaSqに含まれるnvaSq.lowbankを用いる。円筒形状の場合とほぼ同じだが、平板の幅や厚さはsample info fileからではなく、引数として与える。
import nvaSq
d90 = nvaSq.lowbank("./", "10708.rn", "SiO2glass2.in", "90_10steps", , width = 1.4, thickness = 0.1, thickness_cell = 0.2, thickness_std = 0.1 ))
nvaSqの実行例¶
下記の例では、ターミナル上でipythonを起動し、実行コマンドを入力している。
neutron-otomo:sio2 otomo$ ipython --pylab
Python 2.7.6 (default, Sep 9 2014, 15:04:36)
Type "copyright", "credits" or "license" for more information.
IPython 2.4.1 -- An enhanced Interactive Python.
? -> Introduction and overview of IPython's features.
%quickref -> Quick reference.
help -> Python's own help system.
object? -> Details about 'object', use 'object??' for extra details.
Using matplotlib backend: MacOSX
In [1]: import nvaSq
In [2]: d90=nvaSq.basic("./","10708.rn","SiO2glass2.in","90_1steps")
** Read run numbers
No of runs: 4
sample 10708
empty 10704
vanadium 10706
cell none
*** Sample info read from ./SiO2glass2.in
*** Sample cross sections
[['Si', 0.415071, 0.004, 0.0951, 0.3333], ['O', 0.5805, 0.0, 0.0001056, 0.6667]]
*** Standard Sample cross sections
[['V', -0.03824, 5.08, 2.82, 1.0]]
** Read Raw data from ElementContainerArray
##Pix No.=1/1##
** x-axis changed to Wavelength, Lambda
** Normalizaiton with proton numbers
sample: 2145921053.0 emtpy: 2126837868.0 vanadium : 3920793193.0
** Empty background subtraction
Keys: lamda,Intensity,Error
** V-rod peak cut
** Set ttheta so as to be the average of merged pixels
** Attenuation factor calculation of cylindrical geometry
** Apply atten factor to sample & cell
Keys: lamda,Intensity,Error
** Cell subtraction
** Absolute correction of Vanadium Rod
Keys: lamda,Intensity,Error
** Normalization with no. of sample atom
no. of sample atom : 0.0293857872
** Multiple scattering correction of cylidrical geometry
Keys: lamda,Intensity,Error
** Incoherent correction
Keys: lamda,Intensity,Error
** Noramalization with <b>^2
<b>^2 : 0.276005771432 <b^2> : 0.282086968224
** x-axis changed to Momentum transfer (Q)
--- Save Z-Rietveld foramt text data ---
file name: ./10708_sq_90_1steps_72deg.histogramIgor
--- Save S(Q) text data ---
file name: ./10708sq_90_1steps_72deg_0
--- Save const-dQ S(Q) text data ---
file name: ./10708_sq_90_1steps.txt
In [3]:
- 説明:
ipythonの起動
nvaSqのインポート(pythonのコマンドとして読み込み)
データとして、"90_1steps"をしており、90度バンクデータのピクセルすべてを1本にマージしたデータを用いて、補正を実行。
計算結果は、d90として出力され、pythonでの処理に引き続き使用できる。
- テキストデータとして、下記が出力されている。
Z-Rietveld用:10708_sq_90_1steps_72deg.histogramIgor
Qの刻みがdQ/Q = constのS(Q)データ : 10708sq_90_1steps_72deg_0
Qの刻みがdQ = constのS(Q)データ : 10708_sq_90_1steps.txt
処理が終了されると、補正状況を示すプロットが出力される。 また、#10708cor_90_1steps_72deg_0.png、として自動的に保存される。

- 説明:
monitorで規格化
自己吸収係数
バナジウムから計算されるI0(l)・DW・h(l)
多重散乱や非干渉性散乱断面積
多重散乱や非干渉性散乱断面積を引いて得られた干渉性散乱断面積図中の数値は、<bcoh>2
S(Q)
nvaSqにより返り値として出力されたデータの中身¶
出力データは、Manyo-libのElementContainerArrayという形式である。Arrayとして複数のデータ(ElementContainer)を1個にまとめて持つことができる。 ElementContainer同士の四則演算や誤差伝搬が実装されており、また、下記のように、解析に使うさまざまなデータを持つことができる。
In [3]: d90(0).Dump(5)
*** header object start
***Int4Map***
Index Key Value
***DoubleMap***
Index Key Value
0 Lsintheta 9637.98
1 ttheta 1.26883
2 L1 15000
3 L2 1260.94
4 theta 0.634416
5 no_atom 0.0293858
6 <b>^2 0.276006
7 <b^2> 0.282087
***StringMap***
Index Key Value
***Int4VectorMap***
Index Key Size Value
0 MergedPixels 12168 [200012,200013,200014,200015,200016,200017,200018,200019,200020,200021,200022,200023,200024,200025,200026,200027,200028,200031,200032,200033,200034 ......]
***DoubleVectorMap***
Index Key Size Value
0 PixelPosition 3 [ 1203.89,0,375 ]
***StringVectorMap***
Index Key Size Value
*** header object end
The number of vectors is 18
x key = Q
y key = S(Q)
e key = Error
Index Key Size Unit Values
0 Assc 4487 [ 0.844659,0.844658,0.844658,0.844658,0.844658 ,..... ]
1 CohXS 4487 1E-24cm^2 [ 0.278946,0.275308,0.277595,0.278205,0.277521 ,..... ]
2 CohXSError 4487 None [ 0.00130739,0.00129089,0.00129746,0.00129987,0.00129719 ,..... ]
3 DOmega 4487 None [ 0.00331635,0.00335419,0.00335927,0.00335014,0.00334782 ,..... ]
4 D_VALUE 4488 None [ 0.0916142,0.0917064,0.0917986,0.0918909,0.0919834 ,..... ]
5 Empty_proton 4487 counts [ 2.32874e-07,2.49593e-07,2.73549e-07,2.59691e-07,2.60767e-07 ,..... ]
6 Error 4487 (None/None) [ 0.00473681,0.00467703,0.00470084,0.00470957,0.00469987 ,..... ]
7 I(Q)_atoms 4487 None [ 0.311944,0.308306,0.310593,0.311203,0.310519 ,..... ]
8 I(Q)_cell 4487 counts [ 3.04001e-05,3.03884e-05,3.06601e-05,3.06369e-05,3.05484e-05 ,..... ]
9 I(Q)_empty 4487 counts [ 2.56777e-05,2.56678e-05,2.58973e-05,2.58777e-05,2.58029e-05 ,..... ]
10 I(Q)_proton 4487 counts [ 2.59106e-05,2.59174e-05,2.61709e-05,2.61374e-05,2.60637e-05 ,..... ]
11 IncoXS 4487 1E-24cm^2 [ 0.000106093,0.000106093,0.000106093,0.000106093,0.000106093 ,..... ]
12 MultipleSc 4487 1E-24cm^2 [ 0.0328919,0.0328919,0.0328918,0.0328918,0.0328918 ,..... ]
13 Q 4487 angstrom^-1 [ 68.5486,68.4797,68.4109,68.3422,68.2735 ,..... ]
14 S(Q) 4487 None [ 0.988622,0.975441,0.983726,0.985936,0.983458 ,..... ]
15 TOF 4488 microsecond [ 446.398,446.847,447.297,447.747,448.197 ,..... ]
16 Vanadium_proton 4487 counts [ 3.4671e-05,3.50801e-05,3.51563e-05,3.50471e-05,3.50235e-05 ,..... ]
17 lamda 4487 angstrom [ 0.108655,0.108765,0.108874,0.108984,0.109093 ,..... ]
- 説明:
Assc : 自己吸収係数。SiO2ガラスロッドが試料なので、AcscやAccは計算されない。計算値。
DOmega : バナジウムから計算されるI0(l)・DW・h(l)。実測値
Empty_proton : emptyデータをプロトン数で規格化したデータ。実測値
Vanadium_proton : vanadiumデータをプロトン数で規格化したデータ。実測値
I(Q)_proton : sampleデータをプロトン数で規格化したデータ
I(Q)_empty : I(Q)_protonからemptyを差し引いたデータ
I(Q)_cell : I(Q)_emptyから、自己吸収係数を補正したのちにcellを差し引いたデータ
I(Q)_atoms : I(Q)_cellをDOmega(I0(l)・DW・h(l))で割り、原子数で規格化したデータ
MultipleSc : 多重散乱。計算値
IncoXS : 非干渉性散乱断面積。テーブル値からの計算値
CohXS : 補正で求められた干渉性散乱断面積。I(Q)_atoms - IncoXS - MultipleSc
S(Q) : (CohXS - <b^2>) / <b>2 + 1
nvaSqにより返り値として出力されたデータのプロット¶
NVAPlot.pyを使ってプロットする。
In [4]: from NVAPlot import NVAPlot as nvp
In [5]: figure()
Out[5]: <matplotlib.figure.Figure at 0x103fe3f50>
In [6]: nvp(d90(0),yax="CohXS",err=0)
- 説明:
NVAPlotを読み込み。nvpという名前で使えるように指定している。
プロットするwindowを開く
d90(0)、つまりd90に入っている最初のElementContainerの、CohXSを、err無しでプロットする。
- yaxで表示するデータを指定する。
指定できるキーワードは、Assc、Acsc、Acc、DOmega、MultipleSc、IncoXS、CohXS、S(Q)など
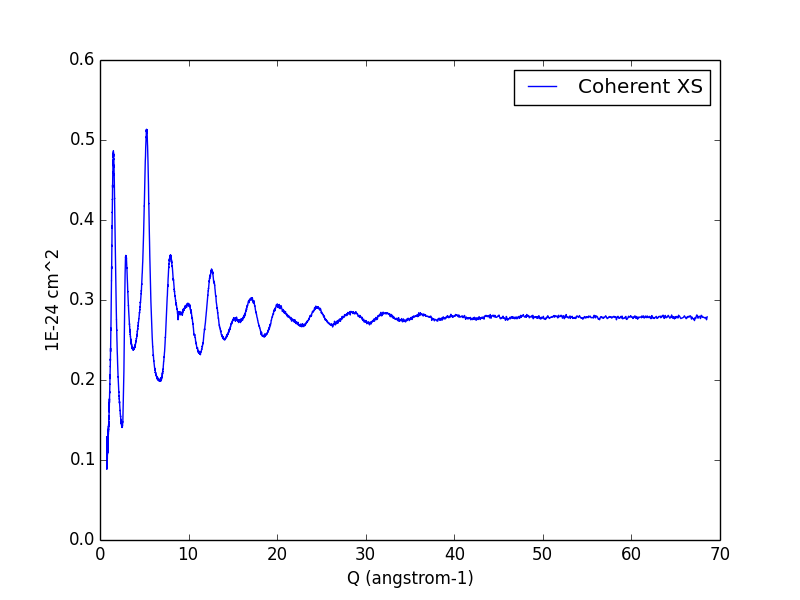
In [37]: nvp(d90(0),yax="S(Q)")
Ltotal(L1+L2): 16260.9443294 , scattering angle(2theta): 72.6987226881
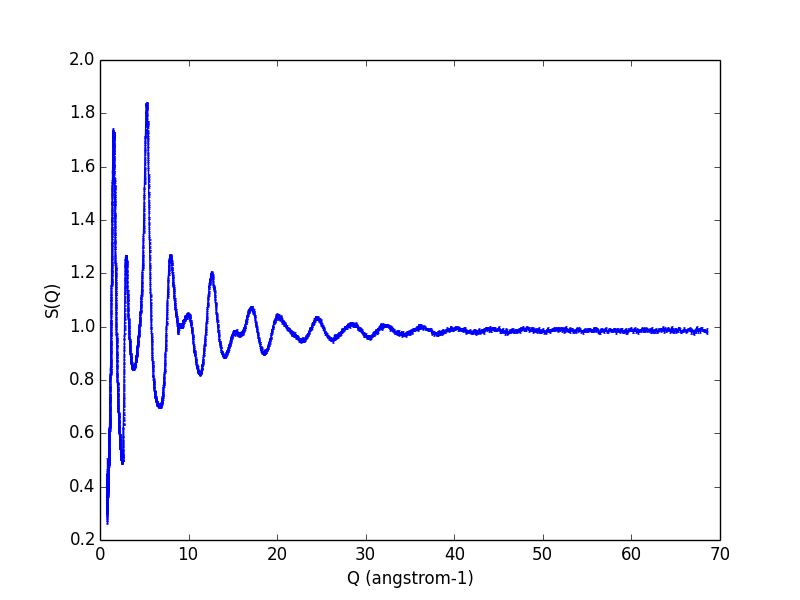
- 説明:
エラーバー付きでS(Q)をプロット。